Reinhart Koselleck-Projekt für Wissenschaftler der Freien Universität Berlin
Deutsche Forschungsgemeinschaft fördert Biochemiker Markus Wahl mit einer Million Euro
Nr. 028/2020 vom 07.02.2020
Die Deutsche Forschungsgemeinschaft (DFG) stellt dem Biochemiker Markus Wahl von der Freien Universität Berlin im Rahmen eines Reinhart Koselleck-Projektes Mittel im Umfang von einer Millionen Euro zur Verfügung. Mit dem Programm gewährt die DFG Wissenschaftlerinnen und Wissenschaftlern, die sich über besondere wissenschaftliche Leistungen ausgewiesenen haben, finanziellen Freiraum für in hohem Maße innovative Forschung. Prof. Wahl leitet seit 2009 die Arbeitsgruppe Strukturbiochemie am Institut für Chemie und Biochemie im Fachbereich Biologie, Chemie, Pharmazie der Freien Universität. Seit 2017 ist er darüber hinaus Leiter der gemeinsamen Forschergruppe Makromolekulare Kristallographie des Helmholtz-Zentrums Berlin und der Freien Universität. Er will die Mittel in den kommenden fünf Jahren dazu verwenden, den Prozess der Transkription in Bakterien besser zu verstehen.
Die Transkription ist der erste Schritt der sogenannten Genexpression in lebenden Zellen. Dabei werden Abschriften von Bereichen des Erbmaterials DNA in Form von RNA-Molekülen erzeugt. Diese RNA-Moleküle dienen unter anderem als Baupläne für die Herstellung von Proteinen. Die Transkription wird von einem komplexen Protein-Enzym vorgenommen, der RNA-Polymerase. Ein Transkriptionszyklus kann in drei Phasen unterteilt werden. Während der Initiation erkennt die RNA-Polymerase sogenannte Promotoren auf der DNA, an denen die Transkription gestartet wird. Während der zweiten Phase, der Elongation, liest die RNA-Polymerase die folgenden Bereiche der DNA ab und baut schrittweise ein RNA-Molekül auf. Die Termination als dritte Phase dient der geordneten Beendigung eines Transkriptionsprozesses und setzt das RNA-Produkt frei. In allen Phasen der Transkription wird die Aktivität der RNA-Polymerase über eine große Zahl von weiteren Proteinen reguliert, sogenannten Transkriptionsfaktoren. Solche Regulationsmechanismen stehen im Zentrum der Forschung von Markus Wahl und seiner Arbeitsgruppe. Neben grundlagenwissenschaftlichen Erkenntnissen könnten die erwarteten Ergebnisse auch die Entwicklung neuer Antibiotika befeuern, die auf die Transkriptionsmaschinerie in Bakterien abzielen.
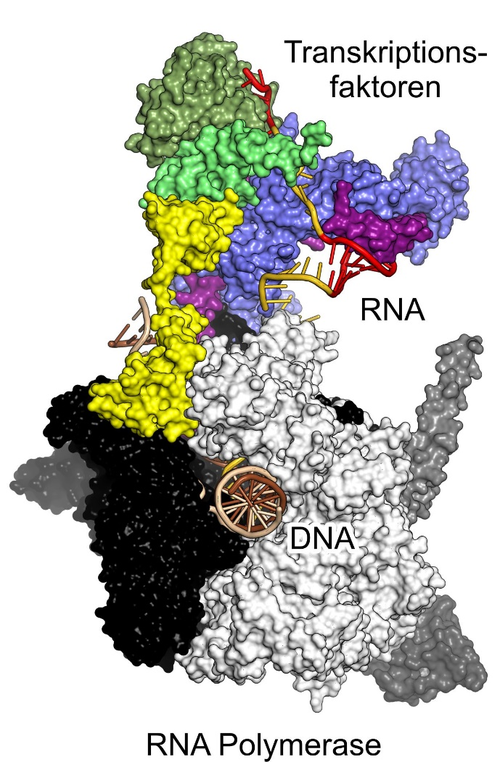
Um molekulare Mechanismen, die der Transkription zugrunde liegen, besser zu verstehen, nutzt die Arbeitsgruppe von Markus Wahl unter anderem leistungsstarke Methoden, die es erlauben, die dreidimensionalen Strukturen biologischer Makromoleküle und ihrer Komplexe im atomaren Detail aufzuklären (siehe Abbildung). Neben der makromolekularen Röntgenkristallographie, für die sich an drei Strahlrohren des Beschleunigerrings BESSY II des Helmholtz-Zentrums Berlin exzellente Voraussetzungen bieten, stehen den Wissenschaftlerinnen und Wissenschaftlern in Berlin von Mitte 2020 an zwei neue Hochleistungs-Elektronenmikroskope zur Verfügung. Die Aufstellung dieser Geräte wurde durch einen erfolgreichen, gemeinsamen Großgeräteantrag der Freien Universität Berlin und der Charité – Universitätsmedizin Berlin, unterstützt durch die Humboldt-Universität zu Berlin und durch Mittel des Landes Berlin im Jahr 2017 ermöglicht. Die Installation des ersten Gerätes auf dem Campus der Freien Universität in Dahlem wurde im Dezember 2019 abgeschlossen. Mitarbeiter der Arbeitsgruppe Wahl konnten mithilfe dieses Mikroskops bereits erste neue 3D-Modelle biomakromolekularer Komplexe erstellen. Der Biochemiker ist von der nun in Berlin zur Verfügung stehenden Infrastruktur für die strukturbiochemische Forschung begeistert: „Die wissenschaftlichen Fragen, die wir in unserer Forschung angehen, lassen sich oftmals nicht mit einer einzigen Methodik erschöpfend beantworten“, betont Markus Wahl. Mit der Installation der neuen Elektronenmikroskope, der Röntgenkristallographie-Strahlrohre an BESSY II, der Hochfeld-NMR Spektrometer am Leibniz-Forschungsinstitut für Molekulare Pharmakologie und höchstauflösenden Lichtmikroskopen an mehreren lokalen Institutionen biete die Berliner Forschungslandschaft nun „ideale Voraussetzungen, um Struktur und Dynamik biologischer Makromoleküle im Detail zu studieren. Die großzügige Bereitstellung von Fördermitteln durch die DFG erlaubt es uns, diese hochmoderne Infrastruktur in den kommenden Jahren effektiv zu nutzen.“
Originalpublikation
Krupp F, Said N, Huang YH, Loll B, Bürger J, Mielke T, Spahn CMT, Wahl MC (2019). Structural basis for the action of an all-purpose transcription anti-termination factor. Mol Cell 74, 143–157.
Pressefoto
3D-Strukturmodell eines Transkriptionskomplexes
Bildquelle: Krupp F, Said N, Huang YH, Loll B, Bürger J, Mielke T, Spahn CMT, Wahl MC (2019). Structural basis for the action of an all-purpose transcription anti-termination factor. Mol Cell 74, 143–157.
Das Foto steht Medien zum Download zur Verfügung und ist bei Verwendung im Kontext der Pressemitteilung und bei Angabe der Quelle honorarfrei.
Weitere Informationen
Deutsche Forschungsgemeinschaft, Reinhart Koselleck-Projekte
https://www.dfg.de/foerderung/programme/einzelfoerderung/reinhart_koselleck_projekte
Kontakt
Prof. Markus C. Wahl, PhD, Fachbereich Biologie, Chemie, Pharmazie der Freien Universität Berlin, E-Mail: markus.wahl@fu-berlin.de
https://www.bcp.fu-berlin.de/en/chemie/biochemie/research-groups/wahl-group/index.html