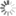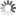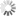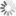„Stoppt den Geisterzug!“ – Wie Bakterien vermeiden, unerwünschte RNA zu produzieren
Science-Publikation mit Beteiligung von Wissenschaftlerinnen und Wissenschaftlern der Freien Universität Berlin
Nr. 235/2020 vom 03.12.2020
Ein wichtiger Prozess bei der Genexpression in Bakterien scheint anders zu verlaufen, als bisher in Lehrbüchern dargestellt. Zu diesem Ergebnis kommt ein internationales Team von Wissenschaftlerinnen und Wissenschaftlern unter der Leitung des Arbeitskreises Strukturbiochemie an der Freien Universität Berlin. Die Arbeiten betreffen unter anderem die Frage, weshalb sich pathogene Bakterien als schädlich für uns erweisen, ohne sich selbst zu schädigen. Die Studie wurde in der Zeitschrift Science veröffentlicht (https://doi.org/10.1126/science.abd1673).
Alle Lebewesen auf der Erde verwenden RNA-Moleküle als Abschriften genetischer Anweisungen auf der DNA, um Proteine zu produzieren, die zelluläre Prozesse vermitteln. Diese RNA-Moleküle werden von RNA-Polymerasen, also komplexen und leistungsstarken molekularen Maschinen, im Vorgang der sogenannten Transkription synthetisiert. Die Transkription ist ein fließbandartiger Prozess: Während eines jeden Arbeitszyklus greift die RNA-Polymerase einen Baustein der DNA-Kette, findet den dazu passenden RNA-Baustein und verknüpft ihn mit der wachsenden RNA-Kette. „Die Polymerase kann unermüdlich RNA-Ketten von bis zu einer Million Bausteinen bilden. Sie ‚erkennt‘ jedoch nicht, ob das von ihr hergestellte RNA-Molekül für die Zellen kritisch, nutzlos oder sogar schädlich ist“, sagt Prof. Dr. Markus Wahl, einer der korrespondierenden Autoren der Studie und Experte für Strukturbiologie an der Freien Universität.
Wenn RNA-Polymerasen nutzlose oder schädliche DNA-Sequenzen transkribieren, anstatt sich auf die DNA-Sequenzen zu konzentrieren, die für die Zelle zu einem bestimmten Zeitpunkt am wichtigsten sind, treffen sie häufig nicht auf Stoppsignale in der DNA, die normalerweise die legitimen Transkriptionsvorgänge beenden. Daher müssen RNA-Polymerasen, die sich auf solchen Abwegen befinden, von bestimmten Proteinen abgeschaltet werden, sogenannten Terminationfaktoren. Diese Terminationsfaktoren führen die Ablösung der RNA-Polymerase von ihrer DNA-Matrize und ihrem RNA-Produkt herbei; der sogenannte Transkriptionskomplex wird aufgelöst.
Das bakterielle Rho-Protein war der erste identifizierte Transkriptionsterminationsfaktor. Rho ist ein sogenanntes Motorprotein, das auf RNA-Molekülen entlangwandern kann und mechanische Kräfte ausübt, die viel stärker sind als die von RNA-Polymerasen ausgeübten Kräfte. Diese und andere Beobachtungen führten zu der vorherrschenden Hypothese, dass das Rho-Protein eine bestimmte Erkennungssequenz auf der RNA bindet und dann mit seinem leistungsstarken Motor die Polymerase einholt und die RNA-Kette aus dem Transkriptionskomplex herauszieht. „Dieses Modell konnte jedoch nie erklären, wie das Rho-Protein die Synthese von RNAs beenden kann, denen solche Eintrittssequenzen fehlen“, sagt Prof. Dr. Irina Artsimovitch, Biochemikerin an der Ohio State University in Columbus, USA, eine weitere leitende Wissenschaftlerin der Studie. „Dazu gehören RNA-Abschriften von Virulenzgenen, deren Expression für Bakterien zu kostspielig ist, außer während einer erfolgreichen Infektion eines Wirtsorganismus.“ Die Fähigkeit des Rho-Proteins, die Transkription abzuschalten, ist somit entscheidend dafür, dass Bakterien erfolgreich in Ruhestadien überleben können, bis sich die Möglichkeit zu einem Angriff auf einen Wirt bietet. Um jedoch ein so genaues Timing zu erreichen, muss das Protein die Transkription sehr schnell stoppen. Das machte es bislang schwierig, Rho experimentell bei seiner Tätigkeit zu beobachten.
Ein Team von Berliner Wissenschaftlerinnen und Wissenschaftlern hat nun zusammen mit Kolleginnen und Kollegen von der Ohio State University, des Centre for DNA Fingerprinting and Diagnostics in Hyderabad, Indien, und der Universität Turku, Finnland, Beobachtungen gemacht, die das obige Lehrbuchmodell in Frage stellen. Dr. Nelly Said und Dr. Tarek Hilal aus der AG Strukturbiochemie der Freien Universität Berlin gelang es, einen Transkriptionskomplex im Prozess der durch das Rho-Protein vermittelten Termination herzustellen und molekulare 3D-Strukturen des Komplexes mit dem Verfahren der kryogenen Einzelpartikel-Elektronenmikroskopie (Kryo-EM) bei hoher Auflösung aufzuklären. Sie ermittelten die Strukturen von sieben Unterzuständen, die anscheinend aufeinanderfolgende Schritte bei der Termination darstellen: angefangen bei der Rho-Rekrutierung bis hin zur Inaktivierung der RNA-Polymerase (Abbildung). „Kryo-EM ist in einzigartiger Weise in der Lage, ein Ensemble selbst instabiler intermediärer biomolekularer Strukturen zu erfassen. Sie ist daher eine ideale Methode, um dynamische Prozesse wie die schnelle aber immer noch stark regulierte Termination der Transkription zu untersuchen“, erklären Nelly Said und Tarek Hilal.
Den Biochemikerinnen und Biochemikern zufolge zeigen die ermittelten Strukturen, dass das Rho-Protein nicht so funktioniert, wie lange Zeit angenommen. Anstatt anfänglich an RNA zu binden, dockt Rho zuerst an die Polymerase und zwei andere Proteine, genannt NusA und NusG. Anstatt an RNA zu ziehen, drückt Rho auf die RNA-Polymerase und bewirkt viele Umlagerungen im Enzym. Die Polymerase wird so gezwungen, ihren Griff auf die DNA zu lockern. Das Protein bewirkt auch, dass die RNA sich aus dem aktiven Zentrum der RNA-Polymerase herausbewegt, wodurch die RNA-Verlängerung effektiv gestoppt und somit die Transkriptionsmaschinerie abgeschaltet wird. „Am bemerkenswertesten ist jedoch, dass Rho diese Wirkung entfaltet, ohne seine starke motorische Aktivität einzusetzen“, sagt Markus Wahl.
Während Kryo-EM die verschiedenen Konformationen eines biomolekularen Komplexes aufdecken kann, kann sie die genauen funktionellen Bedeutungen dieser Zustände nicht direkt erklären. Daher verwendete das Team komplementäre biochemische und genetische Experimente, um die Vorhersagen des alternativen Modells vollständig zu validieren. Die Ergebnisse zeigen, dass die Rho-vermittelte Termination eine molekulare Teamarbeit ist, bei der Nus-Proteine sicherstellen, dass Rho nur bei Bedarf aktiv wird, und dass die RNA-Polymerase, egal wie schnell sie arbeitet, Rho gewissermaßen nicht entkommen kann, da Rho auf dem Transkriptionskomplex „mitfährt“. „Im Nachhinein sind diese Ergebnisse keineswegs überraschend“, erklärt Irina Artsimovitch. „Indem Rho von Anfang an per Anhalter auf der RNA-Polymerase mitfährt und nicht versucht, später aufzuholen, kann Rho sicherstellen, dass nur gewünschte RNAs hergestellt werden.“
„Aufgrund der roboterhaften Natur der RNA-Polymerase stellt die unerwünschte Transkription ein Problem für alle Lebensformen, einschließlich dem Menschen, dar. Es ist daher wahrscheinlich, dass in allen Lebewesen ähnliche Mechanismen für die Qualitätskontrolle bei der Transkription existieren“, konstatiert Markus Wahl.
Abbildung
Kryo-EM-Strukturen, die das Rho-Protein auf dem Weg zur Termination eines Transkriptionskomplexes zeigen.
Bildquelle: Markus Wahl, Freie Universität Berlin
Die Abbildung steht Journalistinnen und Journalisten zum Download zur Verfügung und ist im Kontext der Pressemitteilung und Nennung der Quelle honorarfrei.
Weitere Informationen
Onlinepublikation
Said N, Hilal T, Sunday ND, Khatri A, Bürger J, Mielke T, Belogurov GA, Loll B, Sen R, Artsimovitch I, Wahl MC (2020) Steps toward translocation-independent RNA polymerase inactivation by terminator ATPase ρ. https://doi.org/10.1126/science.abd1673
Kontakt
Prof. Markus C. Wahl, PhD, Freie Universität Berlin, Institut für Chemie und Biochemie, Laboratory of Structural Biochemistry, https://www.bcp.fu-berlin.de/en/chemie/biochemie/research-groups/wahl-group, E-Mail: markus.wahl@fu-berlin.de