Ein „molekularer Raufbold“ recycelt RNA-Polymerase
Drei Forschungsgruppen klären einen neuartigen molekularen Mechanismus für die effiziente Genexpression in pathogenen Bakterien auf / Bild zum Download verfügbar
Nr. 256/2020 vom 18.12.2020
Ein Motorprotein mit der Bezeichnung HelD entfernt die RNA-Polymerase, also das zentrale Enzym bei Transkriptionsvorgängen, von der DNA-Matrize und ermöglicht somit die Produktion weiterer genetischer Botschaften. Zu diesem Ergebnis kommen drei internationale Forschungsteams, darunter ein an der Freien Universität Berlin koordiniertes Konsortium. Die Arbeiten decken molekulare Mechanismen auf, die für das effiziente Wachstum pathogener Bakterien wichtig sind, und darüber hinaus zur Fähigkeit der Bakterien beitragen, dem Immunsystem durch Eintritt in ruhende Zustände zu entkommen. Die Erkenntnisse wurden in der Zeitschrift Nature Communications veröffentlicht (https://doi.org/10.1038/s41467-020-20157-5; https://doi.org/10.1038/s41467-020-20158-4; https://doi.org/10.1038/s41467-020-20159-3).
Alles bekannte Leben hängt vom Fluss der genetischen Information von DNA über RNA zu Proteinen ab. In allen Lebensformen wird der erste Schritt dieses Genexpressionsprozesses, die Transkription von DNA in RNA, von einem komplexen Enzym ausgeführt, das als RNA-Polymerase bezeichnet wird.
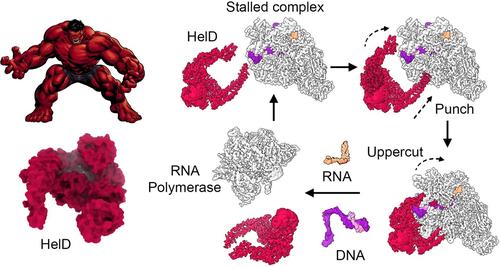
Als einzellige Organismen sind Bakterien immer wieder veränderten Umgebungsbedingungen ausgesetzt, beispielsweise wenn sie ihre Wirte infizieren. Um zu überleben und sich zu vermehren, müssen sie schnell auf diese Umgebungsänderungen reagieren, indem sie ihre Genexpression anpassen. Bakterien begegnen dieser Herausforderung in erster Linie durch eine schnelle Anpassung ihrer Transkription, also der Produktion von RNA, etwa für die Proteinbiosynthese. Zu diesem Zweck muss die RNA-Polymerase optimal funktionieren und die Gene auf der DNA wiederholt transkribieren können. „Die RNA-Polymerase kommt während der Transkription jedoch häufig ins Stocken oder bleibt nach einem Transkriptionsvorgangs auf der DNA hängen, sodass sie nicht weiter transkribieren oder die DNA entlassen kann“, erklärt Prof. Dr. Markus Wahl von der Freien Universität. Derart blockierte RNA-Polymerasen unterminieren dem Biochemiker zufolge die fortgesetzte Genexpression und gefährden die Integrität des gesamten Genoms, da sie Hindernisse für die molekulare Maschinerie darstellen, die die DNA ständig dupliziert. Bisher war in vielen Fällen unklar, wie derartige Blockaden der RNA-Polymerase aufgelöst werden können.
Drei Forschungsteams haben nun aufgeklärt, wie einige Bakterien dieses grundlegende Problem lösen. „Die Bakterien greifen dabei auf ein Molekül namens HelD zurück. HelD besitzt kraftvolle ‚Arme‘, mit denen es an einen muskulösen Comic-Helden erinnert“, sagt Markus Wahl (siehe Abbildung). Das Molekül benutzt diese Arme, um tief in die RNA-Polymerase hineinzugreifen, die Polymerase weit zu öffnen und alle gebundenen Nukleinsäuren zu entfernen. Um diesen erstaunlichen Mechanismus für das Recycling der RNA-Polymerase aufzuklären, verwendeten die drei Forschungsgruppen die sogenannte kryogene Einzelpartikel-Elektronenmikroskopie, einen leistungsstarken Ansatz, für den 2017 der Nobelpreis für Chemie verliehen wurde. Mithilfe der Methode können die 3D-Strukturen von makromolekularen Komplexen bei hoher Auflösung bestimmt werden.
Ein australisches Team unter der Leitung von Prof. Dr. Peter Lewis von der University of Newcastle und Dr. Gökhan Tolun von der University of Wollongong untersuchte den HelD-Komplex der RNA-Polymerase aus einem wichtigen Modellbakterium, Bacillus subtilis, das eng mit gefährlichen menschlichen Krankheitserregern verwandt ist, etwa Bacillus anthracis, dem Erreger von Milzbrand, oder Clostridium difficile, das tödliche pseudomembranöse Colitis und Durchfall verursacht. Die australische Gruppe klärte auch die erste Struktur der Bacillus subtilis RNA-Polymerase im Transkriptionsprozess auf. „Zusammen veranschaulichen diese Strukturen direkt die durch HelD verursachte, dramatische Öffnung der Polmerase“, sagt Prof. Lewis.
Ein tschechisches Team unter der Leitung von Dr. Libor Krasny und Dr. Jan Dohnalek von der Akademie der Wissenschaften der Tschechischen Republik entdeckte eine Variante des gleichen Mechanismus bei Mykobakterien, die Tuberkulose und andere verheerende Krankheiten verursachen. „Das HelD-Molekül in Mykobakterien ist mit einem zusätzlichen Arm ausgestattet, der in die RNA-Polymerase hineingreifen kann. Unsere Strukturen zeigen auch, wie HelD die RNA-Polymerase zuerst mit einem Arm erfasst und dann die anderen Arme verwendet, um das Enzym zu öffnen und die Nukleinsäuren zu entfernen“, merkt Dr. Krasny an.
Schließlich klärte ein Konsortium von Forscherinnen und Forschern aus Deutschland, den USA und Finnland unter der Leitung von Prof. Dr. Markus Wahl von der Freien Universität Berlin auf, wie ein bestimmter Baustein der RNA-Polymerase in einigen Bakterien das Protein HelD in seinem Angriff unterstützt. Diese Gruppe lieferte auch Hinweise darauf, dass das HelD-Molekül bei niedriger Energieversorgung der Zellen eine Verpackung von RNA-Polymerase in inaktiver Form herbeiführen kann. „Interessanterweise benötigt das HelD-Protein die universelle Energiewährung von Zellen, ATP, nicht, um die RNA-Polymerase zu öffnen. Stattdessen ist HelD auf ATP angewiesen, um die RNA-Polymerase wieder loszulassen“, erklärt Prof. Wahl.
Originalpublikationen
- Newing T, Oakley AJ, Miller M, Dawson C, Brown SHJ, Bouwer JC, Tolun G, Lewis PJ (2020) Molecular basis for RNA polymerase-dependent transcription complex recycling by the helicase-like motor protein HelD. Nat Commun, https://doi.org/10.1038/s41467-020-20157-5
- Kouba T, Koval T, Sudzinova P, Pospisil J, Brezovska B, Hnilicova J, Sanderova H, Janouskova M, Sikova M, Halada P, Sykora M, Barvik I, Novacek J, Trundova M, Duskova J, Skalova T, Chon U, Murakami KS, Dohnalek J, Krasny L (2020) Mycobacterial HelD is a nucleic acids-clearing factor for RNA polymerase. Nat Commun, https://doi.org/10.1038/s41467-020-20158-4
- Pei HH, Hilal T, Chen Z, Huang YH, Gao Y, Said N, Loll B, Rappsilber J, Belogurov GA, Artsimovitch I, Wahl MC (2020) The δ subunit and NTPase HelD institute a two-pronged mechanism for RNA polymerase recycling. Nat Commun, https://doi.org/10.1038/s41467-020-20159-3
Kontakte
- Peter J. Lewis
School of Environmental and Life Sciences, University of Newcastle, Callaghan, New South Wales 2308, Australia
https://www.newcastle.edu.au/profile/peter-lewis
email: peter.lewis@newcastle.edu.au
Twitter: @uonmolmicro - Gökhan Tolun
Molecular Horizons and School of Chemistry and Molecular Bioscience, University of Wollongong, Keiraville, New South Wales, Australia
https://scholars.uow.edu.au/display/gokhan_tolun
email: gokhan_tolun@uow.edu.au
Twitter: @gokhan_tolun - Libor Krasny
Laboratory of Microbial Genetics and Gene Expression, Institute of Microbiology, Academy of Sciences of the Czech Republic, Prague, Czech Republic
http://www.biomed.cas.cz/mbu/krasny
email: krasny@biomed.cas.cz
Twitter: @Akademie_ved_CR - Jan Dohnalek
Laboratory of Structure and Function of Biomolecules, Institute of Biotechnology, Academy of Sciences of the Czech Republic, Vestec, Czech Republic
https://www.ibt.cas.cz/en/research/laboratory-of-structure-and-function-of-biomolecules
email: jan.dohnalek@ibt.cas.cz
Twitter: @IBT_CAS - Markus C. Wahl
Laboratory of Structural Biochemistry, Institute of Chemistry and Biochemistry, Freie Universität Berlin, Berlin, Germany
https://www.bcp.fu-berlin.de/en/chemie/biochemie/research-groups/wahl-group
email: markus.wahl@fu-berlin.de
Twitter: @FUStructBiochem